Terapie innovative con i super-algoritmi, anche contro il cancro
La rivista Nature Methods ha dedicato la copertina a un nuovo sistema sviluppato da Bruno Correia (Politecnico di Losanna) in collaborazione con Michael Bronstein (USI), per prevedere il "comportamento" delle proteinedi Elisa Buson
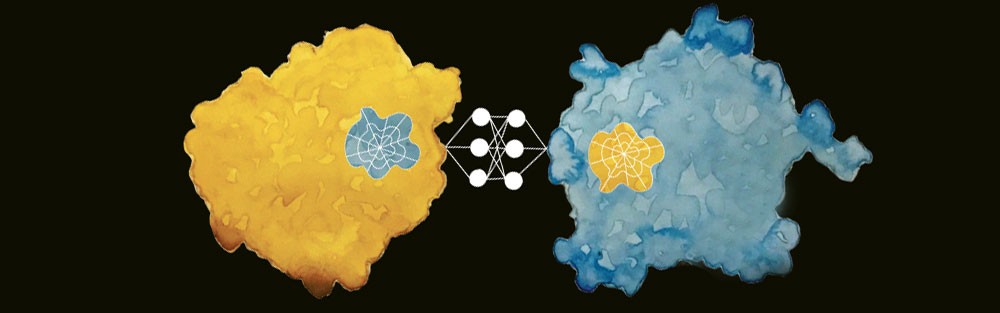
Mai giudicare dalle apparenze. L’abito non fa il monaco, ci hanno sempre detto. Eppure in certi casi la forma è anche sostanza, come nel microscopico mondo delle proteine. Dal loro aspetto è infatti possibile predire quale sarà il loro comportamento: lo dimostra un nuovo sistema di intelligenza artificiale chiamato MaSIF (Molecular Surface Interaction Fingerprinting), che studia la struttura superficiale di questi “mattoni” della vita, la riconosce come una sorta di impronta digitale e la sfrutta per prevedere la loro interazione con le altre molecole biologiche.
Il sistema, che aiuterà a progettare nuovi farmaci e perfino cellule artificiali, è pubblicato sulla rivista Nature Methods (che all’argomento ha dedicato la copertina) da un gruppo internazionale di ricerca coordinato da Bruno Correia del Politecnico di Losanna (EPFL) in collaborazione con Michael Bronstein, professore di informatica all’Università della Svizzera italiana (USI) e all’Imperial College di Londra, nonché capo della ricerca in Graph Learning di Twitter.
«Le proteine possono legarsi ad altre molecole, ad esempio ai recettori delle cellule, come fa una chiave che entra nella serratura: in questo modo inducono una cascata di segnali che porta ad attivare, spegnere o modulare un’attività biologica nella cellula», spiega Bronstein. Come noi impariamo a riconoscere la forma delle chiavi per trovare quella che apre la porta di casa, così i ricercatori hanno insegnato all’intelligenza artificiale a collegare le proprietà geometriche e chimiche delle proteine con le loro capacità di interazione.
Dopo l’addestramento, l’algoritmo è diventato capace di analizzare miliardi di superfici proteiche al secondo. «Ora possiamo studiare anche interazioni multiple tra proteine, confrontando un intero mazzo di chiavi con tante serrature: è come un puzzle tridimensionale, che adesso è possibile risolvere con una velocità fino a 10.000 volte superiore a prima», sottolinea l’esperto.
Questa tecnica aiuterà a capire come interagiscono le circa 20.000 proteine presenti nel corpo umano, ma non solo. Servirà anche a disegnare nuovi farmaci contro il cancro, ad esempio «molecole capaci di legare e inibire il complesso di proteine PD-1/PD-L1 che rende le cellule tumorali invisibili al sistema immunitario», spiega Bronstein. Ma le applicazioni potrebbero andare ben oltre la medicina: si potrebbero disegnare anche proteine da usare come sensori chimici o catalizzatori per velocizzare le reazioni nei processi industriali.
 Michael Bronstein Guarda la gallery (5 foto)
Michael Bronstein Guarda la gallery (5 foto)
A rendere possibili questi progressi è il particolare metodo di apprendimento usato dall’intelligenza artificiale, il cosiddetto “geometric deep learning”, che si è fatto strada negli ultimi cinque anni. La novità sta nell’applicare gli algoritmi che simulano le reti neurali del cervello a dati di natura geometrica, che possono essere strutturati sotto forma di grafi, ovvero reti formate da una serie di punti (chiamati nodi) uniti tra loro da linee (chiamate lati o spigoli del grafo). Qualche esempio pratico? «Oltre allo studio della superficie delle proteine, il geometric deep learning può essere applicato alla computer grafica, per catturare i movimenti in 3D di un attore, oppure alle tecniche di riconoscimento facciale e alla gestione delle informazioni sui social network», afferma Bronstein. Pensiamo per esempio a Facebook: la piattaforma si basa su un grafo sociale, dove gli utenti sono i nodi della rete e i collegamenti fra loro sono i rapporti di amicizia.
Oppure pensiamo a Twitter, dove l’interazione tra utenti e contenuti (espressa dai famosi “like”) determina la selezione dei tweet che compaiono nella timeline in base ai gusti personali. Grazie al geometric deep learning è possibile perfino dare la caccia alle bufale che circolano in rete, come ha intuito proprio Bronstein nel 2017. «Ero ad Harvard per il mio anno sabbatico e in quel periodo si parlava moltissimo di fake news e di come la loro circolazione sia sostanzialmente diversa da quella delle notizie vere», ricorda l’esperto. «Avevamo già ottenuto i primi brevetti sul geometric deep learning e ho pensato che avremmo potuto usarlo in ambito commerciale per addestrare l’intelligenza artificiale a riconoscere le notizie vere e false dal modo in cui si diffondono». Così nel 2018 è nata FABULA AI, una start-up che ha subito solleticato l’interesse di Twitter, tanto da essere acquistata dal colosso di San Francisco nel giro di circa un anno. «Credo che ci sia tantissimo talento e potenziale nell’università, ma c’è una grande distanza tra la pubblicazione di un buon articolo, lo sviluppo di una buona tecnologia e la costruzione di una compagnia di successo», commenta Bronstein. «Ciascuno di questi passi è di un ordine di grandezza più difficile del precedente e richiede un approccio mentale diverso. Una delle difficoltà di portare sul mercato la ricerca accademica è che noi universitari siamo focalizzati sui nostri studi e soltanto dopo cerchiamo di capire come applicarli: è come una soluzione che cerca il problema da risolvere. Per fare una compagnia di successo, bisogna guardare le cose dall’altro lato».
